Proteine beschleunigen bestimmte chemische Reaktionen in Zellen um ein Vielfaches. Den molekularen Mechanismus, mit dem das Protein Ras beschleunigt wird und somit das Zellwachstum bremst, beschreiben Biophysiker der Ruhr-Universität Bochum um Prof. Dr. Klaus Gerwert in der Online-Early Edition der Zeitschrift PNAS.
Mit einer Kombination aus Infrarotspektroskopie und Computersimulationen zeigten sie, dass Ras eine Phosphatkette derart unter Spannung setzt, dass sich eine Phosphatgruppe besonders leicht ablösen lässt - die Bremse für das Zellwachstum. Mutiertes Ras ist an der Tumorbildung beteiligt, weil sich diese Reaktion verlangsamt und die Bremse für das Zellwachstum versagt.
"Unsere Ergebnisse könnten helfen, kleine Moleküle zu entwickeln, die Ras-Proteine auf die richtige Geschwindigkeit zurücksetzen", sagt Prof. Gerwert. "Solche Moleküle wären dann für die molekulare Krebstherapie interessant."
An/Aus: Der Ras-Code
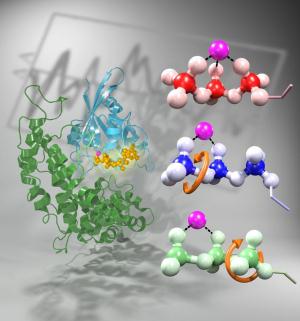
Das Protein Ras schaltet das Zellwachstum ab, indem es eine Phosphatgruppe von dem kleinen gebundenen Guanosintriphosphat, kurz GTP, ablöst. GTP besitzt drei miteinander verkettete Phosphatgruppen. Liegt es in Wasser vor, kann sich die dritte Phosphatgruppe spontan abspalten - auch ohne Hilfe des Proteins Ras. Dieser Prozess ist allerdings sehr langsam. Ras beschleunigt die Abspaltung um fünf Größenordnungen, ein zweites Protein, GAP genannt, um weitere fünf Größenordnungen. Wie es zu dieser Beschleunigung kommt, hat das Bochumer Team nun herausgefunden.
Wie Ras die Phosphatkette spannt
Ras bringt die Kette der drei Phosphatgruppen am GTP in eine bestimmte Form. Es dreht die dritte und zweite Phosphatgruppe so zueinander, dass sich die Kette spannt. "Ähnlich wie man eine Feder in einem Spielzeugauto durch Drehen einer Schraube aufzieht", erklärt Prof. Gerwert. "Ras ist die Schraube, die Phosphatgruppen bilden die Feder." Das Protein GAP spannt die Feder noch weiter, indem es auch die erste Phosphatgruppe gegen die zweite dreht. Damit gelangt das GTP in einen so energiereichen Zustand, dass sich die dritte Phosphatgruppe leicht von der Kette lösen kann - so wie das Spielzeugauto nach Aufziehen der Feder spontan losfährt.
Infrarotspektroskopie: hohe Auflösung, aber nur indirekt zu interpretieren
Die Ergebnisse erzielten die Bochumer Forscher mit der am Lehrstuhl Biophysik entwickelten zeitaufgelösten Fourier-Transform-Infrarot (FTIR)-Spektroskopie. Mit dieser Technik verfolgen die Wissenschaftler Reaktionen und Interaktionen von Proteinen mit hoher räumlicher und zeitlicher Auflösung; sehr viel präziser als mit einem Mikroskop. "Allerdings liefert die Spektroskopie nicht so schöne Bilder wie ein Mikroskop, sondern nur sehr komplexe Infrarotspektren", erklärt PD Dr. Carsten Kötting. "Wie eine Geheimschrift muss man sie dekodieren."
Quantenchemische Simulationen
Dazu simulierte Till Rudack die Proteinreaktionen auf modernsten Computerclustern und berechnete die dazugehörigen Infrarotspektren. Aufgrund des enormen Rechenaufwands lassen sich große Moleküle wie ein komplettes Protein mit dieser sogenannten quantenchemischen Simulation zurzeit nicht zuverlässig beschreiben. Daher beschränkten die Wissenschaftler ihre Analyse auf GTP und den Teil des Ras- bzw. GAP-Proteins, der unmittelbar mit GTP interagiert. Den Rest der Proteine beschrieben sie mit einer weniger aufwendigen Molekulardynamik-Simulation. "Beim Zusammenbringen der verschiedenen Simulationen kann man leicht auf Irrwege geraten", sagt Till Rudack. "Deswegen muss man die Qualität der Ergebnisse prüfen, indem man die simulierten mit den gemessenen Infrarotspektren vergleicht." Stimmen die mit beiden Techniken erhaltenen Spektren überein, kann die Struktur der Proteine bis auf einen millionstel Mikrometer genau bestimmt werden. Das war bei der Bochumer Studie der Fall.
Möglicher Nutzen für die Krebstherapie
Die molekulare Krebstherapie wird bereits erfolgreich bei Erkrankungen wie der chronisch myeloischen Leukämie (CLM) in Form des Medikaments Gleevec angewandt. Ähnlich wirksame Moleküle sind gegen das mutierte Ras-Protein bisher nicht gefunden worden. "Da wir die Reaktionen des Ras-Proteins nun mit deutlich besserer Auflösung untersuchen können, keimt neue Hoffnung auf, das mutierte Molekül mit Wirkstoffen wie Gleevec entschärfen zu können und den richtigen Rhythmus der Zelle wiederherzustellen", so Gerwert.
Zusatzinformationen:
Till Rudack, Fei Xia, Jürgen Schlitter, Carsten Kötting und Klaus Gerwert:
Ras and GTPase-activating protein (GAP) drive GTP into a precatalytic state as revealed by combining FTIR and biomolecular simulations.
In: Proceedings of the National Academy of Sciences; PNAS, online veröffentlicht am 04. September 2012, DOI 10.1073/pnas.1204333109
Quelle: Ruhr-Universität Bochum, RUB
Aktualisiert am 10.09.2012.
Permalink: https://www.internetchemie.info/news/2012/sep12/ras-mechanismus.php
© 1996 - 2026 Internetchemie ChemLin